

全长转录本测序基于单分子实时测序Pacbio Sequel平台,超长读长可获得mRNA全长序列及完整结构信息。克服无参考基因组物种转录本拼接短、信息不完整的难题,实现有参考基因组物种研究可变剪切及融合基因等结构变异。
技术优势
平台优,质量好,性价比高。
基于小而强大的Pacbio Sequel平台,获取mRNA全长序列,
文库构建时无需将转录本打断,信息分析无需组装,精准重构转录组全貌。
科学方案设计:从材料选取,建库测序,到数据分析,每一步都需要科学、缜密的设计,以保障高质量研究成果.
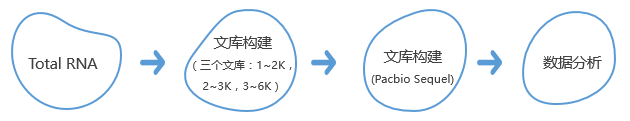
技术路线
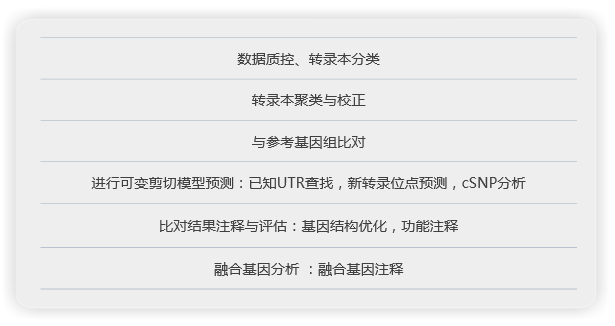
分析内容
样本类型
由于是测全长,所以比二代转录组测序实验对RNA质量的要求更高。
样品量:总RNA>5 ug/文库;总量>15 ug(3个文库);
样品纯度:OD260/280≥1.8,OD260/230≥1.0,260nm处有正常峰值;
RNA 完整性:总RNA的RIN值≥8.0,28S/18S≥1.3;图谱基线无上抬;5S峰正常
Q1:全长转录组测序中,为什么要建3个以上文库?
A:全长转录组采用单分子实时测序技术,通过构建哑铃型文库,以环形方式循环测序。测序时,短片段文库更容易落入零模波导孔。为了避免测序过程中短片段文库偏好性,保证不同长度转录本的覆盖度,因此,会构建3个或3个以上文库。
Q2:全长转录组是否需要打断,拼接?
A:全长转录本是包括从5`末端到3`-poly A tail的,包括mRNA全长序列以及完整结构信息的完整转录本,片段集中分布于1-6kb,sequel平均读长在12kb左右,最长的文库都可以测至少一遍,所以建库前不需要打断,后期分析也无需拼接,得到的就是完整的mRNA。
研究背景
高粱是世界上非常重要的C4作物,也是重要的耐胁迫的谷类,重要的研究非生物胁迫的模式生物。本研究以高粱为研究材料,采用Pacbio的Iso-Seq策略,调研高粱转录组特征,改进高粱基因集注释。
研究结果
1. AS事件分析
本研究发现,有10,053个isoform经历了AS事件,其中只有2,950个isoform和现有的基因模型吻合。Pacbio数据结果证实高粱转录组AS事件比之前的认知更为普遍。同时,文章对在Pacbio中的多isoform的基因做了RT-PCR验证,证实了结果的可靠性。
2. APA事件分析
APA事件是在编码区或3’-UTR区形成isoform,提高转录本的复杂性。由于单分子测序技术利用Oligo dT引物合成cDNA,poly(A)会出现在测序结果中。因此,单分子测序技术是研究APA的有力工具。本文发现在已表达的14,550个基因中,11,013个至少有1个poly(A)位点。
3. 新基因发现
原来的参考基因集有~34,500个基因。在该研究中预测到2171个可能的新基因。为了鉴定这些新基因是否在其他物种中存在,我们使用BLAST中的tblastx和blastx分别比对到收集的植物cDNA序列和Swiss-Prot蛋白数据库,共检测到971个基因。为了证明这些新基因的表达,随机选择7个基因并用RT-PCR验证,证实了新基因的表达和剪切。
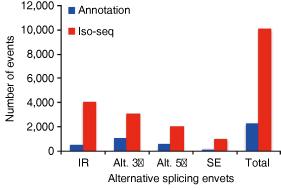
图 Iso-seq和已注释的不同可变剪切类型的比较
参考文献
Abdelghany S E, Hamilton M, Jacobi J L, et al. A survey of the sorghum transcriptome using single-molecule long reads[J]. Nature Communications, 2016, 7:11706.